
基于图像的表型分析(Image-based profiling)是药物发现中的重要工具。表型分析是一种基于图像数据量化细胞变化的策略。高内涵自动成像技术和深度学习算法,特别是计算机视觉技术的进展,可以有效且高效地帮助研究人员确定新的候选药物或靶标所诱导的表型,从而削减开发药物所需的成本和时间。已经采用这种方法取得成功的一个代表案例是近期获得英伟达 5000 万美元私募股权投资的美国 AI 制药上市公司 Recursion Pharmaceuticals。Recursion 的工作流程集成了高内涵、实验室自动化和机器学习方面的创新,使其能够量化和处理数千种表型特征,其中的机器学习算法使他们能够发现原本隐藏的内在关系。
近日,Nature 子刊 Nature Computational Science 发表了浙江大学药学院程翼宇/王毅团队的最新研究成果1,他们通过构建全球首个药物作用下的线粒体动态表型大规模数据集(逾 57 万张单细胞形态图像),研究经 FDA 批准已上市药物的靶点与线粒体表型的相关性,进而发明了一种基于线粒体表型的新药 AI 筛选技术 MitoReID,用于自动学习细胞形态的大规模数据,依据药物高内涵筛选图像推测出其作用机制 MOA,实现了利用细胞筛选模型智能辨识药靶的技术突破。对此技术,Nature 网站 Drug Discovery 专栏以“AI-Recognized Mitochondrial Phenotype Enables Identification of Drug Targets”为题对该论文进行了重点推荐和介绍。同期发表的 Research Briefing 也评价“该方法可通过更好地理解药物作用来改进药物设计”。
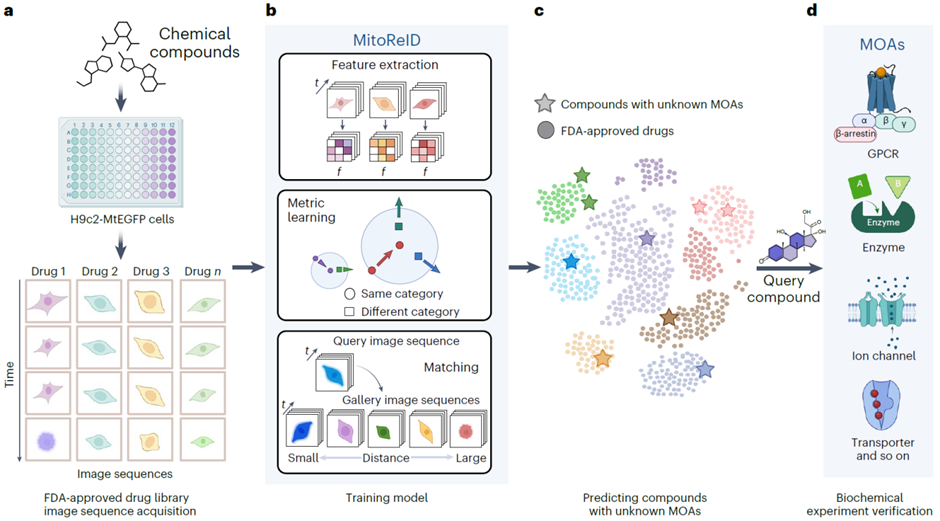
图 1 基于 AI 学习线粒体动态表型的新药 MOA 预测框架,包括四个主要部分:
1. 利用高内涵获取大鼠心肌细胞线粒体经 FDA 药物库处理过的动态图像;
2. 深度学习模型(MitoReID)从各种药物处理后的线粒体图像序列中提取特征,匹配不同的作用机制;
3. MitoReID 从具有未知 MOA 的新化合物中提取特征,并通过与 FDA 药物库的 MOA 进行比较来推断其 MOA;
4. 体外生化实验,以验证未鉴定天然化合物的假设作用机理。
团队首先用绿色荧光蛋白 MtEGFP 和 TMRM 标记 H9c2 心肌细胞线粒体,利用 ImageXpress Micro Confocal 高内涵系统,建立针对线粒体形态、膜电位的表型筛选方法(图 2)。
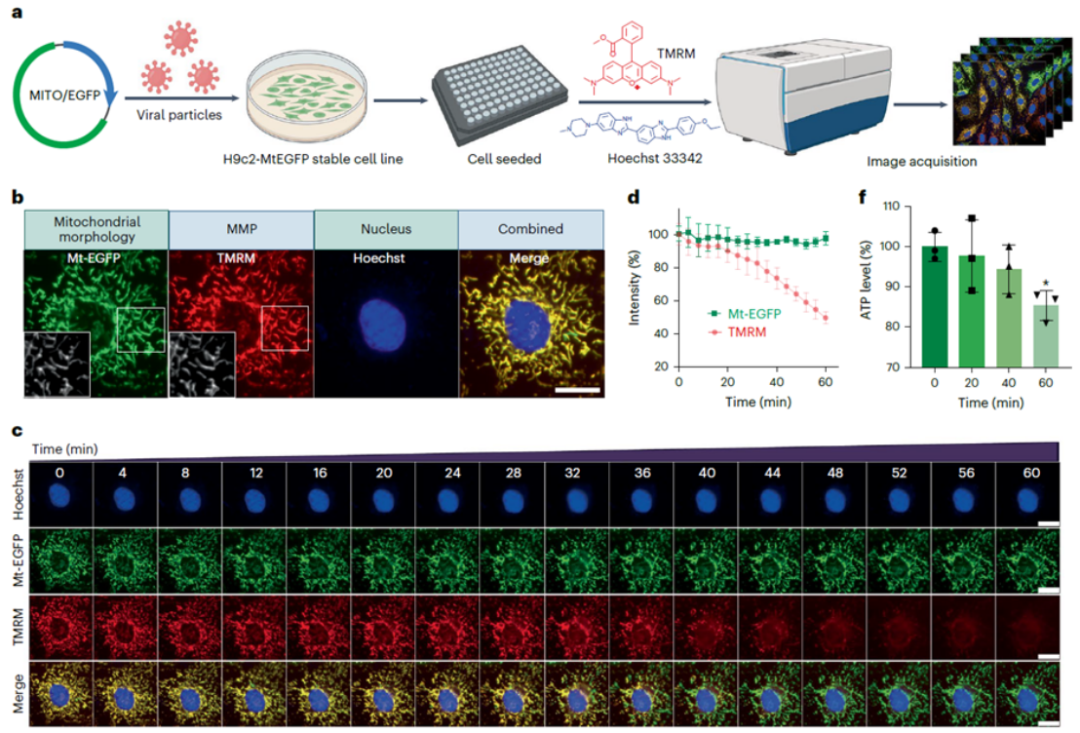
图 2 线粒体形态、膜电位等多维特征表征方法及其时序变化。
通过采集 1068 种 FDA 批准已上市药物作用后的线粒体表型时序动态图像,构建了细胞线粒体动态表型大规模数据集(图 3)。
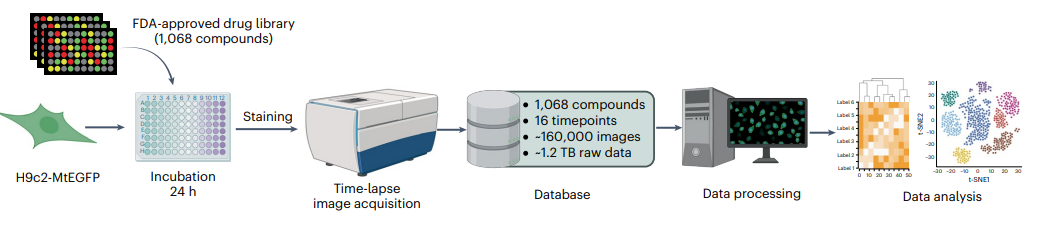
图 3 FDA 药物库处理以后的图像采集和分析流程。
使用 MitoReID 深度学习模型采用重识别框架,通过时间维度增强方法进行自监督预训练,可通过查询匹配新药与训练集中已知靶标药物的线粒体特征相似性来智能推断潜在作用靶标。经过评估,该算法能够将 477 种 FDA 批准药物准确地分类为 38 种已知的 MOA (包括 DMSO)(图 4)。
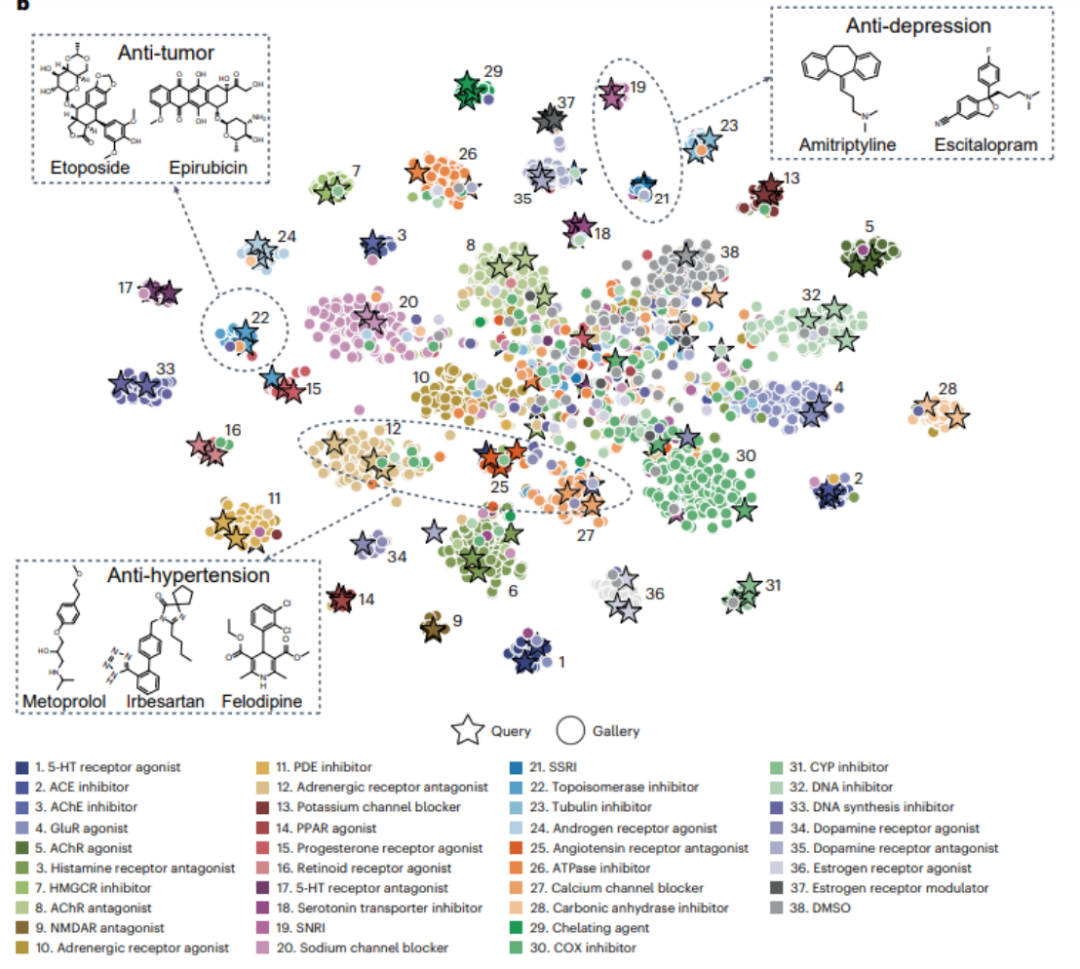
图 4 使用 MitoReID 基于线粒体表型将 477 个药物分类为 38 种作用机制。
实验结果显示,MitoReID 在测试集中的表现优于传统机器学习算法,达到了 76.3% 的 rank-1 准确率和 65.9% 均值平均精度(mAP)。此外,MitoReID 成功识别了 6 种未包含在训练集中的“新药”作用靶标(图 5 显示其中三种)。

图 5 MitoReID 模型用于预测 FDA 批准的药物 MOA。
该模型也被推广用于中药药效物质发现,预测来源于黄芩汤等中药复方/药材中 60 种重要活性成分的潜在作用靶标,发现天然黄烷醇化合物——表儿茶素可能具有抑制环氧合酶(Cyclooxygenase,COX)作用,体外实验进一步验证表儿茶素能直接与 COX-2 蛋白结合,并显著抑制 COX-2 活性。
本研究通过将高内涵采集的大规模时间序列线粒体图像与 MitoReID 的深度学习模型相结合,研究人员成功建立了一种快速、自动化、无偏的预测化学物质 MOA 的方法。通过 AI 技术分析药物对细胞线粒体表型的动态影响,可增进对候选新药作用机制的认识,也能够以更低成本发现中药活性成分的潜在靶标。研究进一步表明,有了高内涵和人工智能驱动的分析平台,科学家们将不再被数据洪流所淹没,而是能够利用他们的创造力和好奇心来发现和开发新的治疗方法。
参考文献
1. Yu, M., Li, W., Yu, Y. et al. Deep learning large-scale drug discovery and repurposing. Nat Comput Sci 4, 600–614 (2024). https://doi.org/10.1038/s43588-024-00679-4.