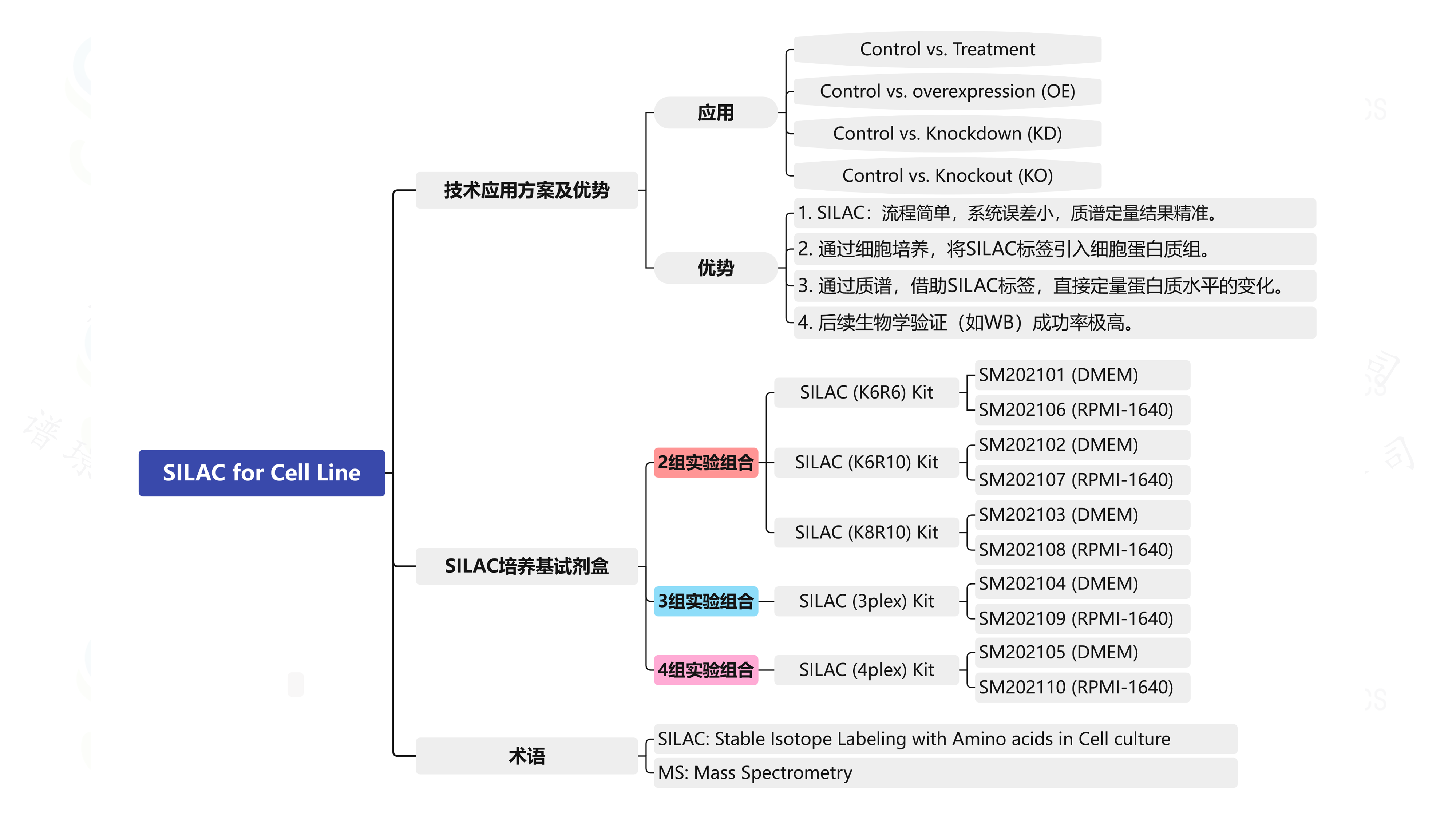
必需氨基酸(如K、R、L、M等)是哺乳动物细胞无法自身合成的氨基酸,必须从食物中摄入。我们使用的哺乳动物细胞系培养的培养基(DMEM,RPMI-1640等)中都加入了“必需氨基酸”。
SILAC (Stable Isotope Labeling with Amino acids in Cell culture) 定量蛋白质组学技术就是利用哺乳动物细胞对“必需氨基酸”依赖的原理,用稳定同位素(2H, 13C, 15N)标记的“必需氨基酸”[如13C6, 15N2-K,缩写为K8,13C6, 15N4-R,缩写为R10] 取代培养基(DMEM, RPMI-1640等)中的普通K(缩写为K0) 和R (缩写为R0),K8比K0在质量上多8 Da (Dalton),R10比R0多10 Da。方便起见,我们将该培养基记为K8R10,普通的培养基我们记为K0R0。
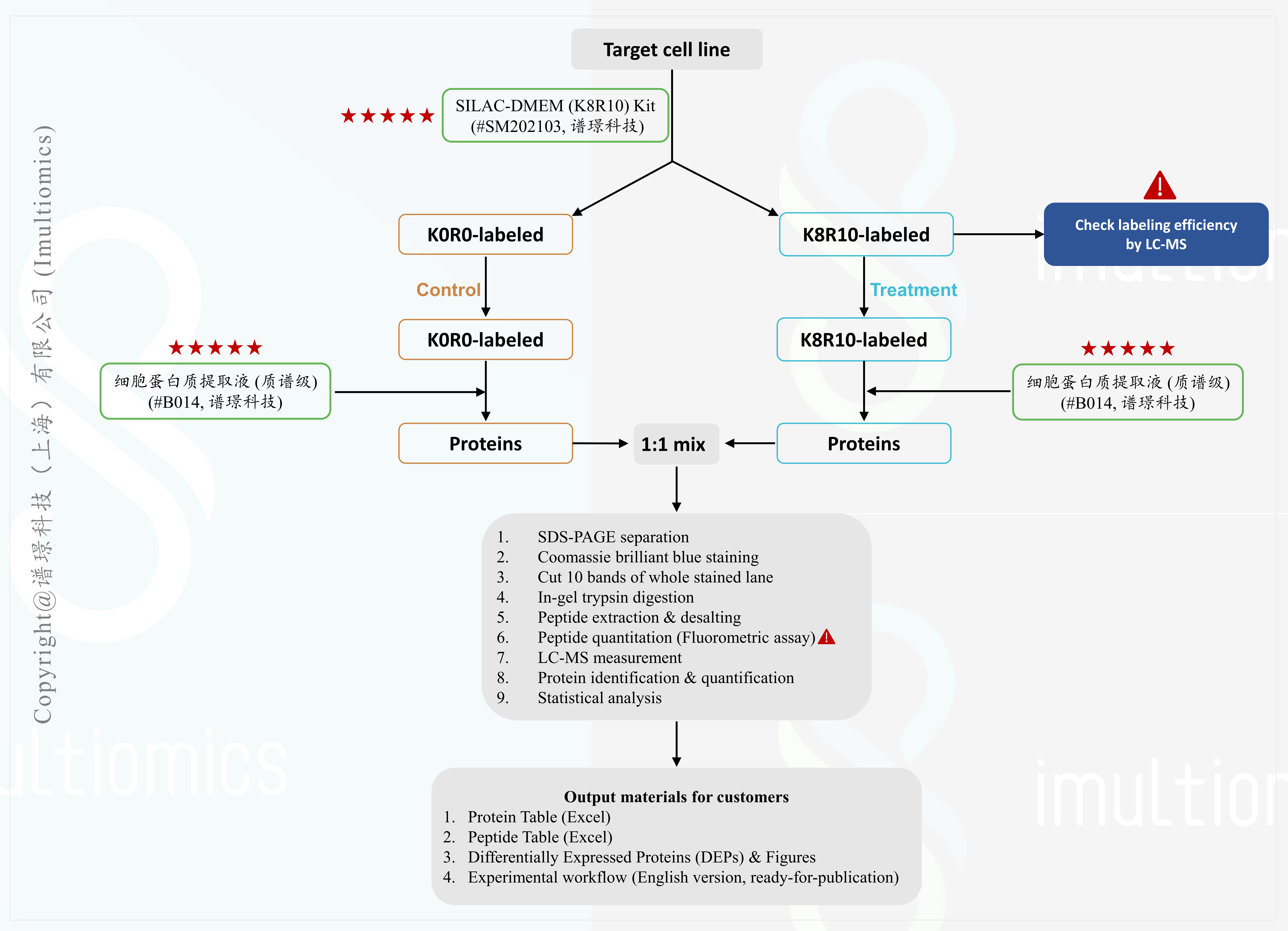
比如我们要比较细胞中某个基因敲除(KO)前后(可应用于各种实验条件,比如KD,过表达、药物处理等等),细胞蛋白质组的差异变化。我们就可以用SILAC技术进行。(1)对KO细胞,我们用K8R10培养基进行培养,则K8和R10通过蛋白质合成就“掺入”到细胞的每个蛋白质分子对应的K和R氨基酸残基上,实现了对细胞蛋白质组(Proteome)的标记,即在每个蛋白质分子上都引入K8和R10的稳定同位素标记;(2)对照细胞,用K0R0培养基培养,合成的蛋白质则是K0和R0标记的。(3)提取2组细胞蛋白,1:1等量混合后,用Trypsin对蛋白进行酶解,Trypsin特异性地从蛋白质的K和R处切割成肽段,生成的肽段几乎都是以K和R结尾的。这样来自两组的蛋白质酶解后生成的肽段是成对的,即XXXXXX-K8 vs. XXXXXX-K0 (8 Da质量差), XXXXXXXXX-R10 vs. XXXXXXXXX-R0 (10 Da质量差 ),每对肽段在一级质谱(MS)上会观察到一对质谱峰(MS Pair),质谱峰的信号强度的相对比值就是该肽段对应蛋白质在2组样品中的表达水平差异。根据SILAC的蛋白质相对定量信息,最终通过统计分析,筛选显著差异表达的蛋白质。
SILAC进行差异蛋白质表达谱筛选是其基本的应用。SILAC技术的高阶应用在分子互作和后修饰(蛋白质-蛋白质互作、RNA-蛋白质互作、小分子-蛋白质互作、DNA-蛋白质互作、蛋白质翻译后修饰)等定量分析领域,定量准确,后续生物学验证阳性率高。
*谱璟科技SILAC-细胞定量质谱分析方案及产品*
谱璟科技基于其技术积累,可开展多种类型的SILAC-细胞定量蛋白质组学实验服务。
谱璟科技有多样化的SILAC产品线,现货供应,可迅速开展实验,缩短实验周期。
谱璟科技具备成熟的细胞生物学平台,可轻松完成对细胞SILAC标记、传代和各种实验条件处理。
谱璟科技具备稳定的质谱技术平台,无缝衔接上游制备的蛋白质样品,轻松开展蛋白质的质谱鉴定、定量、生信分析,快速筛选差异表达的蛋白。
依托我们的分子与细胞技术平台,在步骤4的基础上,对候选差异蛋白进行下游的WB、qPCR验证以及后续更为深入的功能与机理解析服务。
*SILAC-细胞差异表达蛋白质组分析项目DMEO数据*
一、实验设计与流程:
二、DEMO数据:
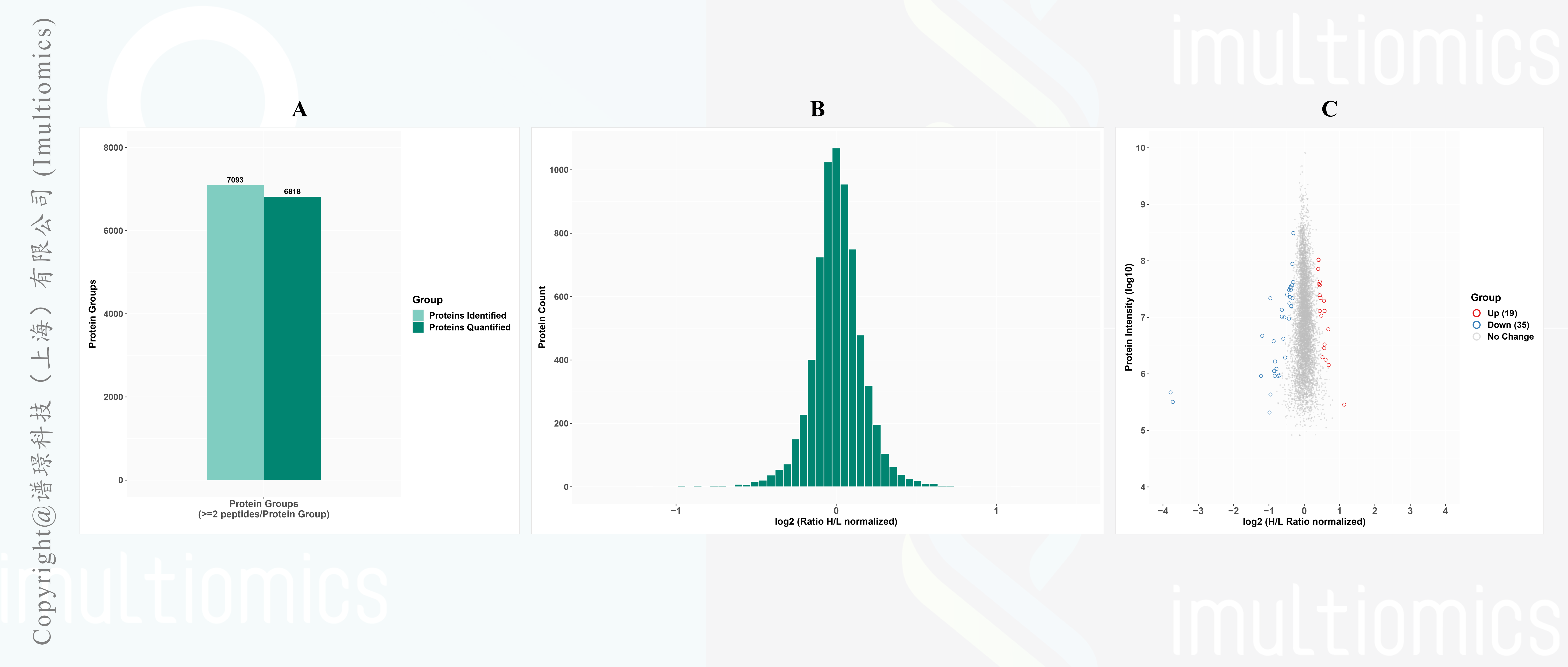
A. 蛋白质鉴定与定量数目统计。共鉴定到>=2条肽段的蛋白质7093种,其中有定量信息的蛋白质为6818种,占比为96.12%。B. 定量蛋白质的log2 (Ratio H/L normalized) 分布图。C. 差异蛋白质筛选。共筛选(Benjamin-Hochberg FDR < 0.05)到上调蛋白19种 (Up)和下调蛋白35种(Down)。
参考文献:
Hoedt E, Zhang G, Neubert TA: Stable isotope labeling by amino acids in cell culture (SILAC) for quantitative proteomics. Advancements of mass spectrometry in biomedical research 2019:531-539.
我司助力的代表性SILAC研究案例:
Cao T, Zhang L, Zhang Y, Yan G, Fang C, Bao H, Lu H: Site-Specific Quantification of Protein Ubiquitination on MS2 Fragment Ion Level via Isobaric Peptide Labeling. Analytical Chemistry 2017, 89(21):11468-11475.
Di Y, Zhang Y, Zhang L, Tao T, Lu H: MdFDIA: A Mass Defect Based Four-Plex Data-Independent Acquisition Strategy for Proteome Quantification. Analytical Chemistry 2017, 89(19):10248-10255.